Kapitel 9
Framtidsutsikt: Metylering
Metylering av DNA är en kemisk modifiering, där en metylgrupp kopplas på en eller flera nukleotider. Metyleringen kvarstår efter replikationen men endast på de ursprungliga DNA-strängarna. Efter replikationen metyleras de nya komplementära DNA-strängarna genom enzymet DNA-metyltransferas i en process som kallas underhållsmetylering.

Figur 10. Illustration av en DNA-molekyl med två metylerade cytosiner. DNA-metylering spelar en viktig roll vid epigenetisk reglering, vilket innebär att DNA modifieras på olika sätt, vilket påverkar genuttrycket. Exempel på en annan epigenetisk modifiering är histonmodifiering.
Hos eukaryota organismer sker metyleringen på kvävebasen cytosin och i sekvenser av cytosin och guanin så kallade GC-sekvenser. Regioner där CG-sekvenser är vanligare kallas CpG-öar och återfinns ofta i närheten av promotorer, den del av DNA där gener aktiveras och RNA-polymeras binder in. DNA-metylering inaktiverar genuttryck därför att transkriptionsfaktorer hindras från att binda in till promotorn och initiera transkription och metylerade CG-områden är lågt transkriberade och på så sätt reglerar metylering genernas aktivering.
Metylering och cancer
Flera typer av cancer uppvisar ett onormalt metyleringsmönster, till exempel kan CpG-öar i promotorregionen till olika tumörsuppressorgener vara hypermetylerade, vilket innebär en nedreglering av tumörsuppressorer. Omvänt kan också promotorer för onkogener vara hypometylerade, vilket driver på tumörutvecklingen.
Aberranta metyleringsmönster är ett verktyg som kan användas i olika kliniska situationer, exempelvis tidig upptäckt av cancer, klassificering, val av behandling samt prognos. En spännande applikation är identifiering av primärtumören hos patienter med cancer utan känd primärtumör med hjälp att klassificeringsmodeller baserade på metyleringsdata.
Hur används metylering i diagnostiken?
I rutindiagnostiken idag används metylering för att identifiera olika undergrupper av hjärntumörer och WHO:s klassificering av CNS-tumörer inkluderar numera även metyleringsanalyser som en del av diagnosen.
Metylering av MGMT-genens promotor används vid val av behandling för glioblastom, den vanligaste och mest aggressiva formen av gliom. Metylering av MGMT-genens promotor kan prediktivt påverka hur väl patienter svarar på behandling med läkemedel.
För utredning av Lynch syndrom används metyleringsanalys av MLH1-genen för en del av utredningen. Vid Lynch syndrom har patienten nedärvda mutationer i någon av generna i DNA-reparationssystemet, DNA mismatch repair, (MLH1, MSH2, MSH6, och PMS2), vilket leder till instabilitet i genomet genom att ovanligt många fel uppstår i replikationen. Detta kan man se genom att analysera sekvensen i så kallade mikrosatelliteter. Mikrosatelliter är områden på DNA med repetitiva sekvenser, exempelvis dinukelotida sekvenser TATATATA eller trinukleotida GCTGCTGCT. Om genen för MLH1 är muterad på den ena allelen inaktiveras genom metylering på den andra allelen driver det på utvecklingen av cancer. Metylering av MLH1 genen förekommer i mikrosatellitinstabil, sporadisk kolorektalcancer men analyseras för att utesluta Lynchsyndrom. Patienter med Lynch syndrom drabbas oftare än andra av kolorektal- och endometriecancer.
Vilket provmaterial?
Metyleringsanalyser kan utföras på alla typer av provmaterial som man kan extrahera DNA ifrån. Det som är begränsande är hur mycket DNA som behövs för respektive analysmetod nedströms.
För de flesta metoder krävs att man gör en modifiering av det extraherade DNA:t innan det kan analyseras. Modifieringen innebär att alla ometylerade cytosin, C, konverteras till uracil, U. Konverteringen görs vanligtvis genom en bisulfitbehandling men även enzymatiska metoder finns numera tillgängliga. Fördelen med de enzymatiska metoderna är att de är mer skonsamma mot DNA och fungerar även då man endast har tillgång till låga koncentrationer av DNA.
Efter konvertering kan man antingen använda sekvenseringsbaserade metoder för att ta reda på vilka C som var metylerade eller ometylerade alternativt riktade PCR- eller array-tekniker.
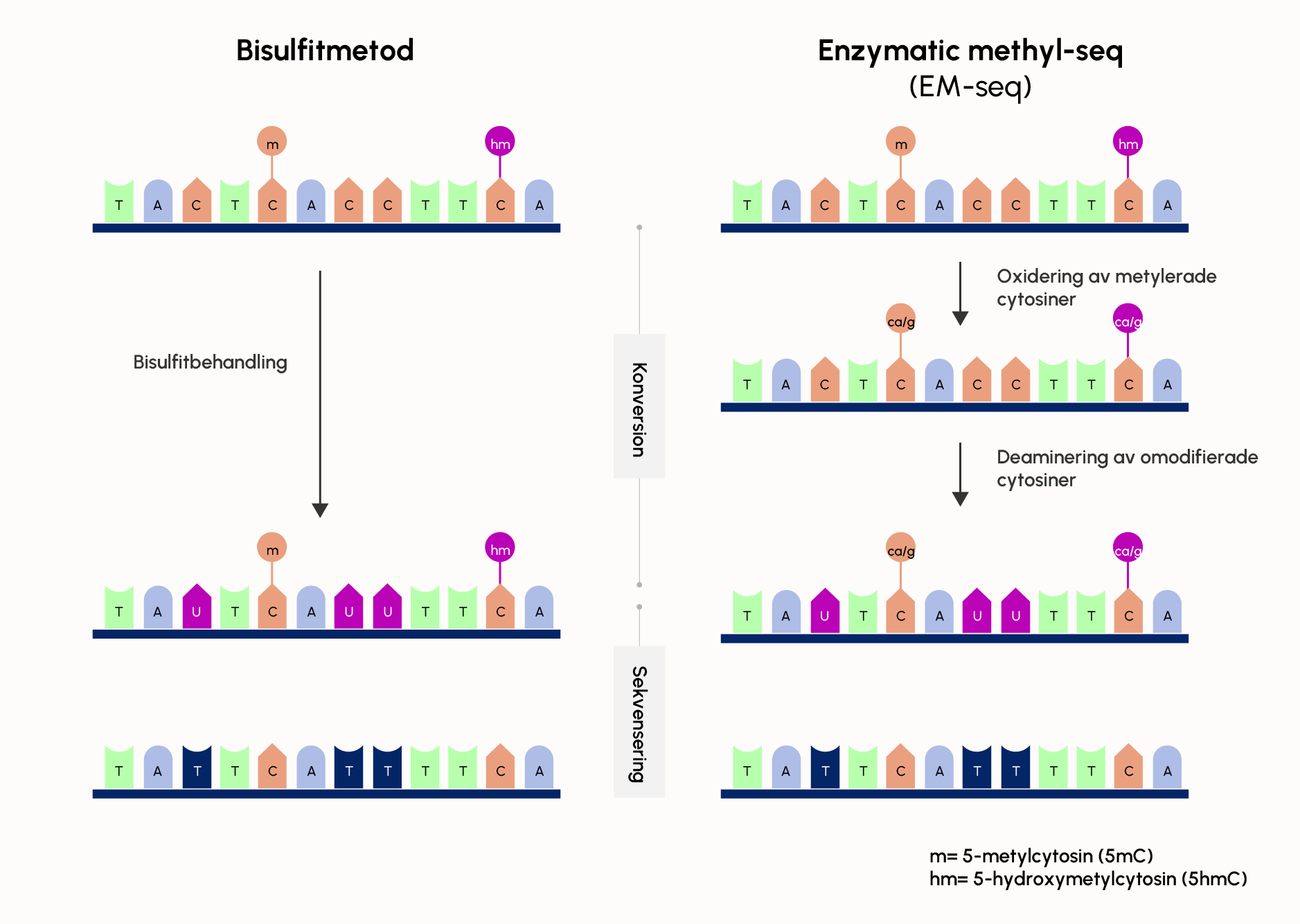
Figur 11. För att kunna analysera metylering måste DNA konverteras, vilket innebär att ometylerade cytosin konverteras till uracil medan metylerade cytosin kvarstår. Konverteringen kan ske kemiskt genom bisulfitbehandling (till vänster) eller enzymatiskt (till höger) Vid bisulfitbehandlingen sker konverteringen i ett steg medan den enzymatiska konverteringen sker i två efterföljande steg.
Referens: Vaisvila R, Ponnaluri VKC, et al. Enzymatic methyl sequencing detects DNA methylation at single-base resolution from picograms of DNA. Genome Res. 2021 Jul;31(7):1280-1289. doi: 10.1101/gr.266551.120.
Vilka metoder kan man använda?
Efter modifieringen av DNA kan man använda samma metoder som används inom molekylärdiagnostiken för övrigt, qPCR, dPCR, array- och sekvenseringsbaserade tekniker.
Valet av metod beror på hur mycket information man vill få ut och vad analysen får kosta. Vill man enbart analysera en specifik gen eller många gener samtidigt? Mängd och kvalitet på DNA som finns tillgängligt och kraven på känslighet styr också metodvalet. Om man bara vill analysera enstaka gener är det oftast mest fördelaktigt att välja qPCR. När man behöver en hög känslighet kan man välja dPCR. För att analysera många gener samtidigt väljer man array- eller sekvenseringsbaserade tekniker. Array-baserade tekniker är etablerade i klinisk rutin och används när man vill undersöka stora delar av genomet och större genetiska varianter. Begränsningen med array-tekniken är att det behövs ganska mycket DNA av hög kvalitet, vilket gör det svårare att använda DNA från formalinfixerad paraffininbäddad vävnad. Med next generation sequencing (NGS) kan man välja att analysera utvalda gener i olika paneler alternativt att analysera hela metylomet, det vill säga metyleringsstatus i hela genomet. NGS-analyser följs av avancerad dataanalys i så kallade bioinformatiska pipelines. NGS finns implementerat i klinisk rutin för genetisk karaktärisering av tumörprover men än så länge inte för metyleringsanalyser. Inom Genomic Medicine Sweden har man utvecklat riktade NGS-paneler för både solida tumörer och hematologiska maligniteter.
För att analysera metylering i klinisk rutin används array-tekniken för att klassificera CNS-tumörer. För metyleringsstatus av MGMT-genen används pyrosekvensering eller qPCR.
Framtida användningsområden för metyleringsanalyser
De senaste åren har mycket forskning initierats för att hitta nya biomarkörer i blodet, så kallade liquid biopsies. Bland annat kan man analysera mutationer i cirkulerande tumör-DNA från plasmaprover och få ut behandlingsstyrande och prognostisk information. Exempelvis för lungcancerpatienter finns det stora möjligheter att ersätta den invasiva vävnadsprovtagningen med ett blodprov.
Liquid biopsies har stor potential och med enzymatisk konvertering och NGS kan man också analysera metyleringsstatus i cirkulerande tumör-DNA. Metoden skulle kunna användas för att detektera cancer i ett blodprov då metyleringsgraden är olika i DNA från cancerpatienter jämfört mot icke-cancer. Då olika typer av vävnader liksom olika cancerformer har distinkta metyleringsprofiler kan man också använda metyleringsanalyser för att fastställa tumörens ursprung. Man kan tänka sig flera olika kliniska tillämpningar för den här metoden exempelvis inom primärvården för patienter som söker för blod i avföringen. Där skulle metoden kunna användas för att urskilja de patienter som ska utredas ytterligare för cancer. Metyleringsanalysen skulle också kunna användas inom det standardiserade vårdförloppet ”Allvarliga ospecifika symptom som kan bero på cancer”.
Författare: Gisela Helenius, docent vid Örebro Universitet, oktober 2025. Läs mer om författaren »
Källor och mer läsning
- Nationellt vårdprogram, Tumörer i hjärna, ryggmärg och dess hinnor, 2025-04-22 Version: 4.1
- Nationellt vårdprogram, Tjock- och ändtarmscancer 2025-05-13 Version: 4.0
- Hassan Ashktorab & Hassan Brim. DNA Methylation and Colorectal Cancer. Curr Colorectal Cancer Rep. 2014 Aug 31;10(4):425–430.
- Central Nervous System Tumors, WHO Classifications on Tumours, 5th Edition, Volume 6, 2021 https://publications.iarc.fr/Book-And-Report-Series/Who-Classification-Of-Tumours/Central-Nervous-System-Tumours-2021
- Duckett D et al. Accurate identification of primary site in tumors of unknown origin (TUO) using DNA methylation. Precision Oncology volume 9, Article number: 8 (2025)
- Qvick A et al. Liquid biopsy as an option for predictive testing and prognosis in patients with lung cancer. Mol Med. 2021 Jul 3;27(1):68.
- Shen SY et al. Sensitive tumour detection and classification using plasma cell-free DNA methylomes. Nature 2018 Nov;563(7732):579-583
- Binliang Wang et al. Circulating tumor DNA methylation: a promising clinical tool for cancer diagnosis and management. Clin Chem Lab Med. 2024 Mar 7;62(11):2111-2127.
SWE-NP-0625-80010


